内蒙古大学 呼和浩特 010000
摘要:近年来,circRNA由于其稳定,保守,分布范围广等优点,成为了表观遗传修饰中倍受瞩目的一员,其对于癌症的发生、发展发挥着至关重要的作用。胃癌作为一种严重威胁人体健康的疾病,它的早期诊断、预后判断、治疗靶点具有很重要的意义。本文将基于GEO数据库,对GSE78092,GSE141977,GSE83521三组芯片数据进行整合分析,最后得出与胃癌具有良好关联性的circRNA,并通过预测其调控的miRNA,进而分析其影响的生物学功能,最后得出该circRNA潜在影响通路和功能,具有高度的临床转化价值。
关键词:胃癌;circRNA;数据库
胃癌是世界范围内最常见的恶性肿瘤之一,发病率居世界第四,肿瘤相关死亡率位居世界第五。[1]我国是胃癌的“重灾区”,发病率、死亡率居高不下。[2]由于我国医疗资源分布不均衡,胃癌早期筛查普及率低,多数患者就诊时已处于进展期,手术治疗难以实现根治,转移率高,预后差。特别是由于饮食结构改变及社会压力增加,近年来胃癌发病呈现出年轻化的倾向,并且青年患者肿瘤具有侵袭性强、体积大、分化程度低等恶性生物学特点胃癌作为具有鲜明地域特色的恶性肿瘤,给患者带来极大的精神和身体负担,严重制约我国经济和社会发展。因此,挖掘具有高度临床价值的早期诊断标志物、预后判断参考值和个体化治疗靶点迫在眉睫。
circRNA的生物学特性如下:(1)稳定性。反向拼接而成的连续、闭合环状结构使circRNA对RNA核酸外切酶的降解不敏感。circRNA稳定存在于多种极端环境,如尿液、胃液中,是临床 非侵袭性诊断方式的适宜标志物。(2)半衰期长。circRNA的半衰期通常大于48h,探测结果稳定,具有参考性。(3)保守性。真核细胞circRNA具有高度保守的位点,极大提升了稳定干扰 疾病相关circRNA表达水平的可行性。所以,凭借结构和功能的独特优势,circRNA在胃癌的临床诊治中具有极大的潜力。
芯片的下载
GEO数据库搜索关键词circRNA 和 gastric cancer 可以得到十八个相关结果。经过初步筛选,得到与本研究相关的数据有以下六组:
GSE152309 五个胃癌患者的新鲜肿瘤组织和相邻非肿瘤组织的RNA测序。
GSE141977 三例胃癌患者的肿瘤组织及癌旁正常组织进行环状RNA表达谱检测。
GSE131414 三例胃癌患者的肿瘤组织及癌旁正常组织环状RNA表达谱检测。
GSE122796 三例胃癌患者的肿瘤组织及癌旁正常组织环状RNA表达谱检测。
GSE83521 六个肿瘤和正常邻近黏膜组织环状RNA表达谱检测。
GSE78092 三例胃癌与邻近的正常黏膜组织相比环状RNA表达谱检测。
表1 circRNA表达谱芯片一般情况
芯片名 | 作者 | 日期 | 平台 |
GSE152309 | Xiaolin W | PubliconJun12,2020 | GPL18573 |
GSE141977 | Nanishi K | PubliconDec14,2019 | GPL21825 |
GSE131414 | Ju S,Shen X | PubliconJun16,2019 | GPL24676 |
GSE122796 | Wang S,Chen J | PubliconNov22,2018 | GPL11154 |
GSE83521 | Zhang Y | PubliconDec14,2017 | GPL19978 |
GSE78092 | Huang YS,Xie N[3] | PubliconFeb20,2016 | GPL21485 |
由于各个芯片中的命名规则不同,最后选择命名规则统一的GSE78092,GSE141977,GSE83521三组胃癌circRNA芯片进行研究。这三组数据,研究者与样本均来自中国,测序数据均为近五年样本,且测序数据均大于3000,符合生物信息学筛选差异化表达circRNA的一般要求。芯片基本信息见表1。
数据的预处理
利用维恩图工具对三组芯片中的交集数据,进行筛选,发现三组数据共有1801组数据相同,取这1801组circRNA的数据研究。由于每个芯片中表达数据处理方式不同,根据归一化处理,将三组数据的表达值进行归一化处理,保证所得结果的合理性。。
三、基于Limma包的差异表达circRNA的筛选
在R.3.5.0环境下,对 GSE78092,GSE141977,GSE83521 胃癌circRNA表达谱芯片数据分别进行Limma包[4]做差异表达分析,过滤条件为校正P<0.05,log2FC绝对值>1,得到GSE78092、GSE100170、GSE83521合并数据的差异表达circRNA。并对差异表达circRNA进行聚类分析热图和火山图的可视化。
可以得出,ASCRP005317,ASCRP005169等circRNA在正常组织中高表达,在胃癌组织中低表达;ASCRP003232,ASCRP004355在正常组织中低表达,在胃癌组织中高表达。从图3可以得出,ASCRP000411,ASCRP004679等circRNA在正常组织中高表达,在胃癌组织中低表达;ASCRP004842,ASCRP000905在正常组织中低表达,在胃癌组织中高表达。可以发现ASCRP000411、ASCRP005317、ASCRP004851、ASCRP002139、ASCRP000430这五个circRNA具有较好的区分度。经过比对,我们可以发现,在两组数据中,ASCPR000430是唯一一个均具有优秀区分度的circRNA,通过查询芯片数据,我们可以挖掘出在胃癌组织和正常组织中,更具有显著差异分布的circRNA为hsa_circ_0009594。
得出circRNA后,我们利用circbank数据库(http://www.circbank.cn/searchMiRNA.html)预测其参与调控的miRNA[5]有以下十五个:hsa-miR-571、hsa-miR-1270、hsa-miR-3192-5p、hsa-miR-377-3p、hsa-miR-4433a-3p、hsa-miR-4722-5p、hsa-miR-4731-5p、hsa-miR-4772-3p、hsa-miR-4776-5p、hsa-miR-5683、hsa-miR-6165、hsa-miR-6856-5p、hsa-miR-6878-3p、hsa-miR-6892-3p、hsa-miR-7974。
四、区分度最佳的circRNA调控基因的GO功能、KEGG通路富集分析[6]
在得出一系列miRNAs后,我们利用 mirdb数据库(http://mirdb.org)[7]预测其gene symbol[8],经过大量数据的分析,我们通过miRDB、miRTarBase、TargetScan三个miRNA数据库进行对比,最后取交集得出了具有可研究性的870个 gene symbol。
然后利用Hiplot数据库
(https://hiplot.com.cn/advance/clusterprofiler-go-kegg),在p阈值0.01,q阈值0.05条件下进行GO功能、KEGG通路富集分析[9]得出以下结果。
图1 GO功能、KEGG通路富集分析图
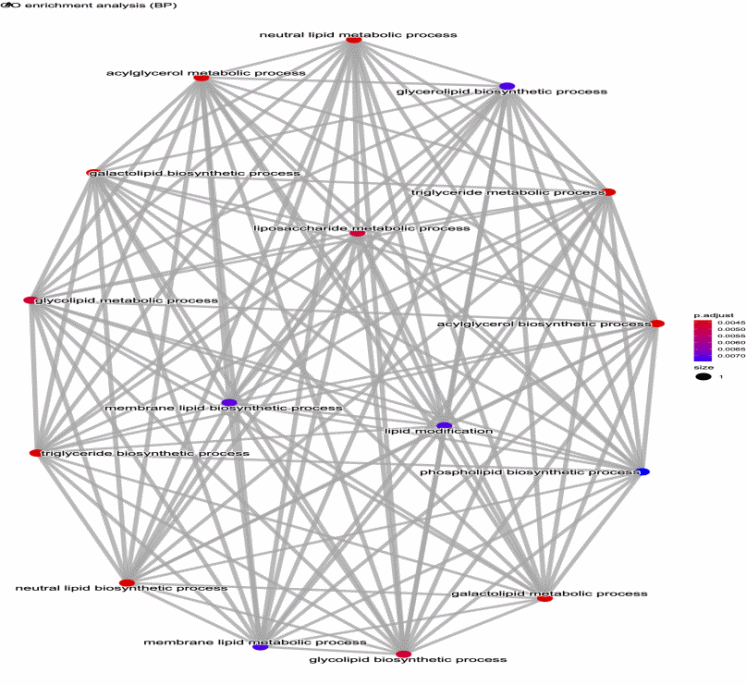
由图可以看出,hsa_circ_0009594在如下GO功能存在富集:糖脂代谢过程、中性脂质合成过程、脂质修饰、翻译监管活性、核酸结合。hsa_circ_0009594在KEGG通路富集层面,在癌症的转录信号通路得到显著富集。
目前的circRNA命名规则不统一,基于GEO数据库进行的分析具有一定的困难。本研究通过分析GSE78092,GSE141977,GSE83521三组芯片数据,分别进行差异化处理取交集分析得出较为良好的区分度circRNA,并对其调控的miRNA进行了预测,并且对其可能影响的通路进行了富集,得出了一些列的结果。由于目前对胃癌中的circRNA的研究较少,本研究所提供的一些数据可以为以后在胃癌中circRNA的探索提供一些思路与证据。
综上所述,本研究基于GEO数据库所下载的胃癌相关的circRNA数据进行了一系列数学和可视化处理,得出了几个具有优秀区分度的环状RNA,并预测其相关的miRNA-DNA,对其影响的生物功能进行了合理的预测。本研究为探索circRNA早期诊断胃癌的初步尝试,为以后的研究提供了具有一定意义的参考,本研究得出的circRNA,可能成为将来胃癌的预测标志物。
1] Cheng J, Guo JM, Xiao BX, et al. PiRNA, the new non-coding RNA, is aberrantly expressed in human cancer cells[J]. Clin Chimica Acta, 2011, 412(17/18):1621-1625.
2] [2] van Cutsem E, Sagaert X, Topal B, et al. Gastric cancer[J].Lancet, 2016, 388(10060):2654-2664.
3] Huang YS, Jie N, Zou KJ, Weng Y. Expression profile of circular RNAs in human gastric cancer tissues. Mol Med Rep 2017 Sep;16(3):2469-2476.
4] Smyth GK.Iimma:linear models for microarray data[J].Bioinformatics & Computational Biology Sollutions Using R & Bioconductor,2011:397-420.
5] Liu M,Wang Q,Shen J,Yang BB,Ding X. Circbank: a comprehensive database for circRNA with standard nomenclature.RNA Biol.2019 Jul
6] Yu G, Wang L, Han Y and He Q*. clusterProfiler: an R package for comparing biological themes among gene clusters. OMICS: A Journal of Integrative Biology. 2012, 16(5):284-287.
7] Weijun Liu and Xiaowei Wang (2019) Prediction of functional microRNA targets by integrative modeling of microRNA binding and target expression data. Genome Biology. 20(1):18.
8] Yuhao Chen and Xiaowei Wang (2020) miRDB: an online database for prediction of functional microRNA targets. Nucleic Acids Research. 48(D1):D127-D131.